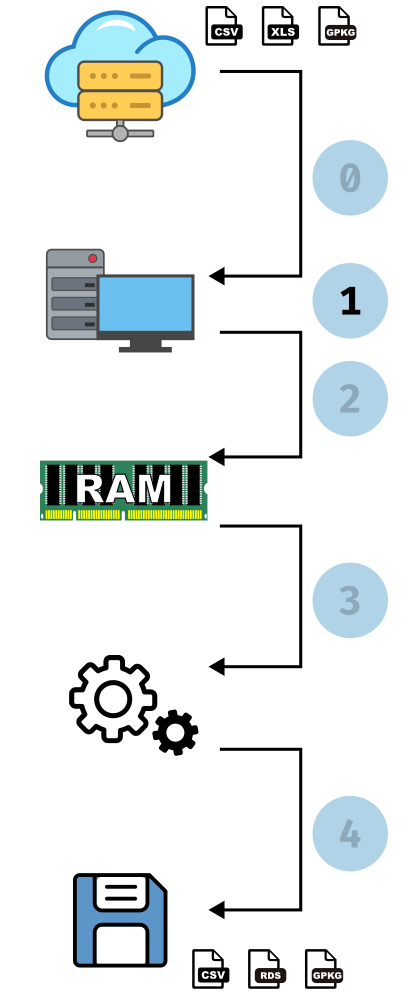
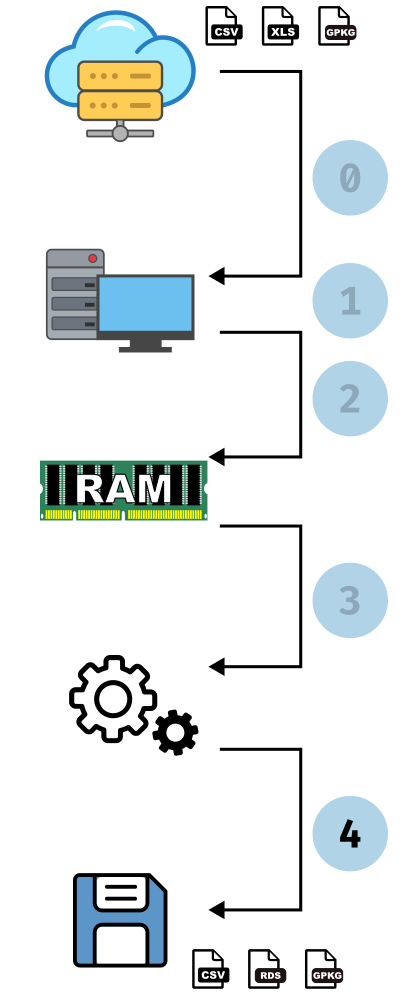
Manipulation de données tabulaires massives
DuckDB, Arrow, Parquet et compagnie
Ré-outiller la chaîne de traitement




Un exemple d’analyse
Analyser le jeu de données des logements ordinaires en 2020 du recensement.
Exercice du jour
Pour chaque commune de France, on cherche à récupérer le nombre de logements, leur distribution selon le type de logement (appartement, maison, etc.) et selon la catégorie de logement (résidence principale, secondaire, logement vacant, etc.).
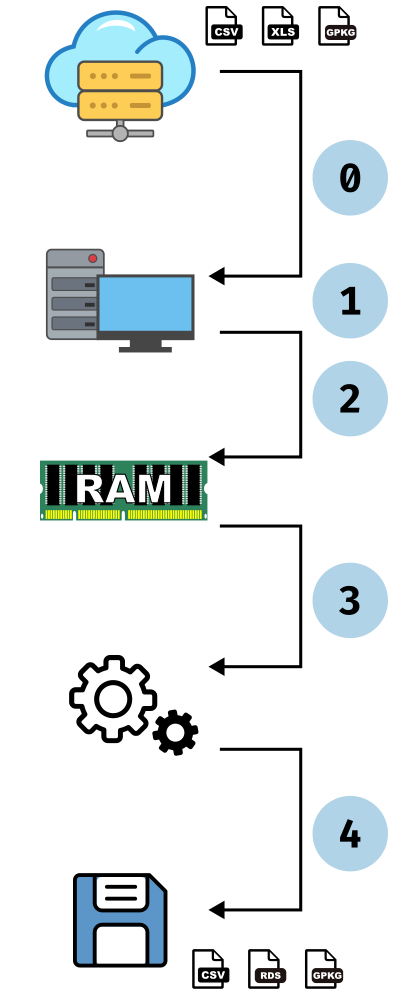
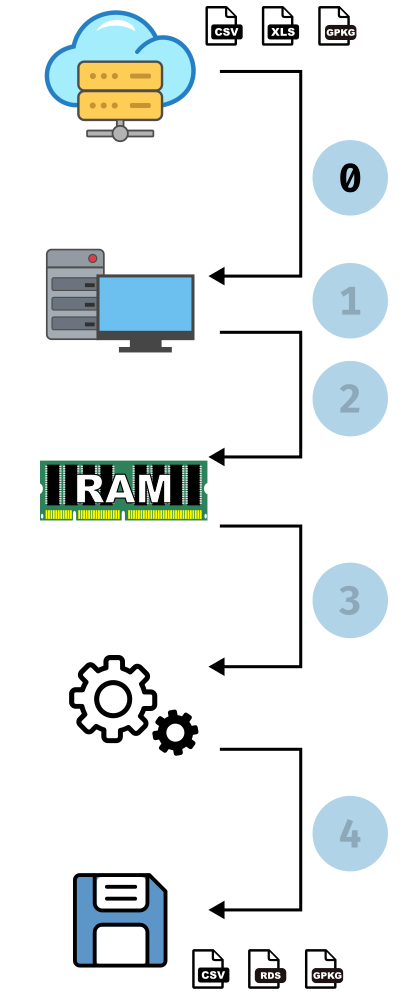
Un exemple d’analyse
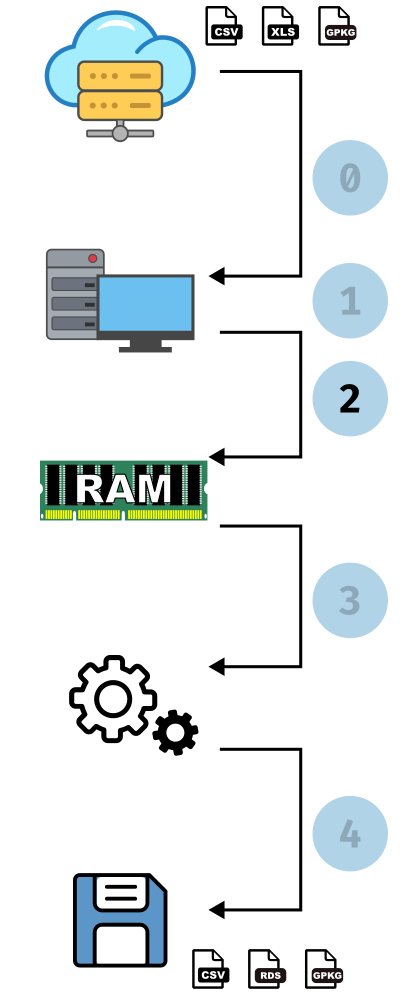
- Téléchargement du fichier
- Extraction de l’archive
- Lecture dans R
- Traitement : récupérer, pour chaque commune, le nombre de logements et le détail de leurs types (maison, appartement, etc.) et de leur catégorie (résidence principale, secondaire, vacant etc.)
- Export du fichier résumé
Un exemple d’analyse
- Téléchargement du fichier
Statistiques
: 58 sec : 372 Mo
Un exemple d’analyse
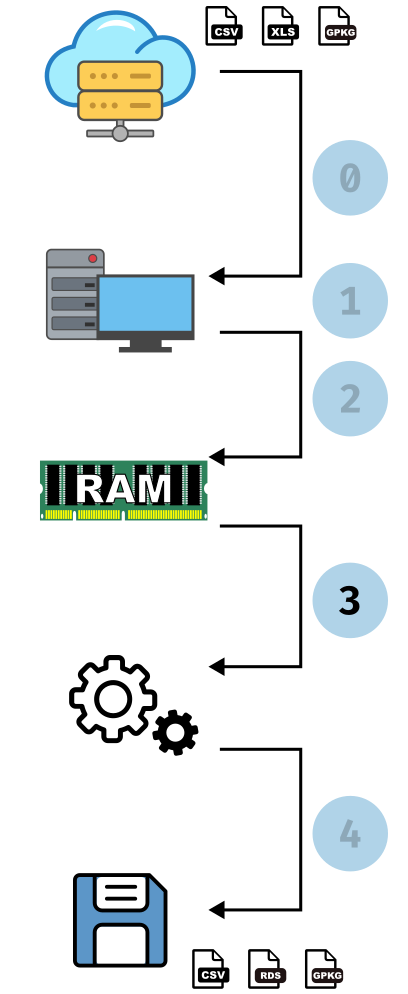
- Lecture dans R
# A data.frame: 25,795,751 × 69
COMMUNE ARM IRIS ACHL AEMM AEMMR AGEMEN8 ANEM ANEMR ASCEN BAIN BATI
<chr> <chr> <chr> <chr> <int> <chr> <chr> <int> <chr> <chr> <chr> <chr>
1 01001 ZZZZZ ZZZZZZZ… A11 1993 8 65 29 4 2 Z Z
2 01001 ZZZZZ ZZZZZZZ… C113 2013 9 40 9 2 2 Z Z
3 01001 ZZZZZ ZZZZZZZ… C113 2013 9 65 9 2 2 Z Z
4 01001 ZZZZZ ZZZZZZZ… A12 1994 8 65 28 4 2 Z Z
5 01001 ZZZZZ ZZZZZZZ… C115 0 0 YY 999 99 Y Z Z
6 01001 ZZZZZ ZZZZZZZ… A12 2021 9 40 1 0 2 Z Z
# ℹ 25,795,745 more rows
# ℹ 57 more variables: CATIRIS <chr>, CATL <chr>, CHAU <chr>, CHFL <chr>,
# CHOS <chr>, CLIM <chr>, CMBL <chr>, CUIS <chr>, DEROU <chr>, DIPLM <chr>,
# EAU <chr>, EGOUL <chr>, ELEC <chr>, EMPLM <chr>, GARL <chr>, HLML <chr>,
# ILETUDM <chr>, ILTM <chr>, IMMIM <chr>, INAIM <chr>, INEEM <chr>,
# INP11M <chr>, INP15M <chr>, INP17M <chr>, INP19M <chr>, INP24M <chr>,
# INP3M <chr>, INP60M <chr>, INP65M <chr>, INP5M <chr>, INP75M <chr>, …
Un exemple d’analyse
- Lecture dans R
# A data.frame: 25,795,751 × 69
COMMUNE ARM IRIS ACHL AEMM AEMMR AGEMEN8 ANEM ANEMR ASCEN BAIN BATI
<chr> <chr> <chr> <chr> <int> <chr> <chr> <int> <chr> <chr> <chr> <chr>
1 01001 ZZZZZ ZZZZZZZ… A11 1993 8 65 29 4 2 Z Z
2 01001 ZZZZZ ZZZZZZZ… C113 2013 9 40 9 2 2 Z Z
3 01001 ZZZZZ ZZZZZZZ… C113 2013 9 65 9 2 2 Z Z
4 01001 ZZZZZ ZZZZZZZ… A12 1994 8 65 28 4 2 Z Z
5 01001 ZZZZZ ZZZZZZZ… C115 0 0 YY 999 99 Y Z Z Statistiques
: 184 sec : ~25 Go en pic (toute la mémoire disponible), ~13 Go après
Un exemple d’analyse
- Traitement
- On réalise un compte par commune des logements selon chaque type et chaque catégorie
- On agrège ce compte par commune en fonction de la catégorie, puis on passe le tableau résultant en format large.
- Idem mais pour les types : agrégation par commune, puis passage en format large.
- On compte le nombre total de logements par commune
- On joint ces trois tableaux

Un exemple d’analyse
- Traitement
'data.frame': 34933 obs. of 12 variables:
$ COMMUNE : chr "24297" "63237" "76090" "19022" ...
$ LOGEMENTS_TOTAL: int 475 355 505 201 769 173 119 166 1363 141 ...
$ CATL_1 : int 387 211 472 124 613 155 72 142 1160 46 ...
$ CATL_3 : int 34 60 8 63 80 7 34 10 2 84 ...
$ CATL_2 : int 1 0 1 0 1 0 1 1 4 9 ...
$ CATL_4 : int 53 84 24 14 75 11 12 13 197 2 ...
$ TYPL_6 : int 1 1 0 0 1 0 0 0 3 0 ...
$ TYPL_3 : int 0 0 0 0 3 0 0 0 1 0 ...
$ TYPL_2 : int 33 18 21 2 11 1 4 19 225 12 ...
$ TYPL_4 : int 0 0 0 0 0 0 0 0 0 0 ...
$ TYPL_5 : int 1 3 1 1 0 0 0 0 0 1 ...
$ TYPL_1 : int 440 333 483 198 754 172 115 147 1134 128 ...Statistiques
: 4.6 sec
Analyse de la chaîne de traitement
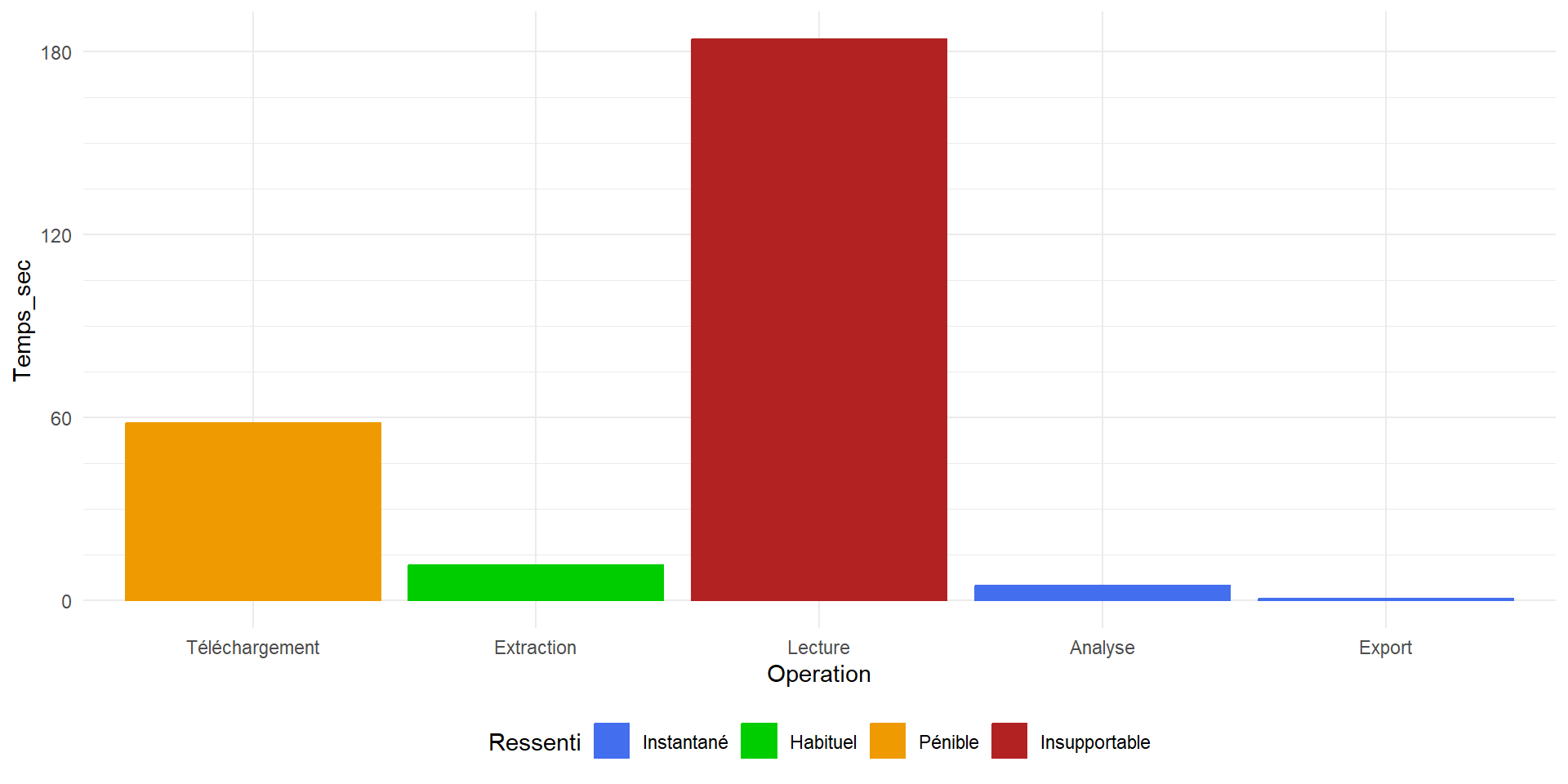
C’est un peu long…
Le tidyverse (et ses amis) à la rescousse !
Le tidyverse (et ses amis) à la rescousse !
- Remplacement du téléchargement par le package
curl: pas de timeout et plus robuste en cas de coupure pour les gros fichiers : - Téléchargement :
download.file()curl::curl_download()
Le tidyverse (et ses amis) à la rescousse !
- Remplacement de l’extraction native par le package
archive: gère plus de formats, plus rapide, des paramètres plus complets - Extraction :
unz()archive::archive_extract()
Le tidyverse (et ses amis) à la rescousse !
- Remplacement de la lecture en data.frame par le package
readr: plus rapide*, plus versatile, génère untibble - Lecture :
read.csv2()readr::read_csv2()
Le tidyverse (et ses amis) à la rescousse !
- Remplacement des analyses
basepar de la syntaxedplyrettidyr: c’est quand même plus explicite1 - Analyse :
aggregate(), reshape(), merge()group_by(),summarise(),pivot_wider()etleft_join()
Le tidyverse (et ses amis) à la rescousse !
- Remplacement de l’export par
readr: on peut enfin ne plus préciser qu’on ne veut pas exporter les numéros de ligne… - Export :
write.csv()readr::write_csv()
Le tidyverse (et ses amis) à la rescousse !
- Téléchargement :
curl::curl_download()
- Extraction :
archive::archive_extract()
- Lecture :
readr::read_csv2()
- Analyse :
group_by(),summarise(),pivot_wider()etleft_join()
- Export :
readr::write_csv()
Adaptation de la chaîne de traitement : 0 & 1
- Téléchargement
- Extraction
Statistiques - Téléchargement
: 57.91 sec : 372 Mo
Statistiques - Extraction
: 5.61 sec : 4.54 Go
Adaptation de la chaîne de traitement : 2. Lecture
Statistiques
: 205 sec : ~22 Go en pic, ~13 Go après
Adaptation de la chaîne de traitement : 3. Analyse
library(dplyr)
library(tidyr)
resume_commune <- rp2020 %>%
group_by(COMMUNE, CATL, TYPL) %>%
summarise(nb_logements = n(),
.groups = "drop")
resume_CATL <- resume_commune %>%
group_by(COMMUNE, CATL) %>%
summarise(nb_logements = sum(nb_logements),
.groups = "drop") %>%
pivot_wider(id_cols = COMMUNE,
names_from = CATL,names_prefix = "CATL_",
values_from = nb_logements,
values_fill = 0)resume_TYPL <- resume_commune %>%
group_by(COMMUNE, TYPL) %>%
summarise(nb_logements = sum(nb_logements),
.groups = "drop") %>%
pivot_wider(id_cols = COMMUNE,
names_from = TYPL, names_prefix = "TYPL_",
values_from = nb_logements,
values_fill = 0)
resume_global <- resume_commune %>%
group_by(COMMUNE) %>%
summarise(LOGEMENTS_TOTAL = sum(nb_logements),
.groups = "drop") %>%
left_join(resume_CATL, by = "COMMUNE") %>%
left_join(resume_TYPL, by = "COMMUNE")Statistiques
: 4.00 sec
Adaptation de la chaîne de traitement : 4. Export
Statistiques
: 0.7 sec : 1.2* Mo
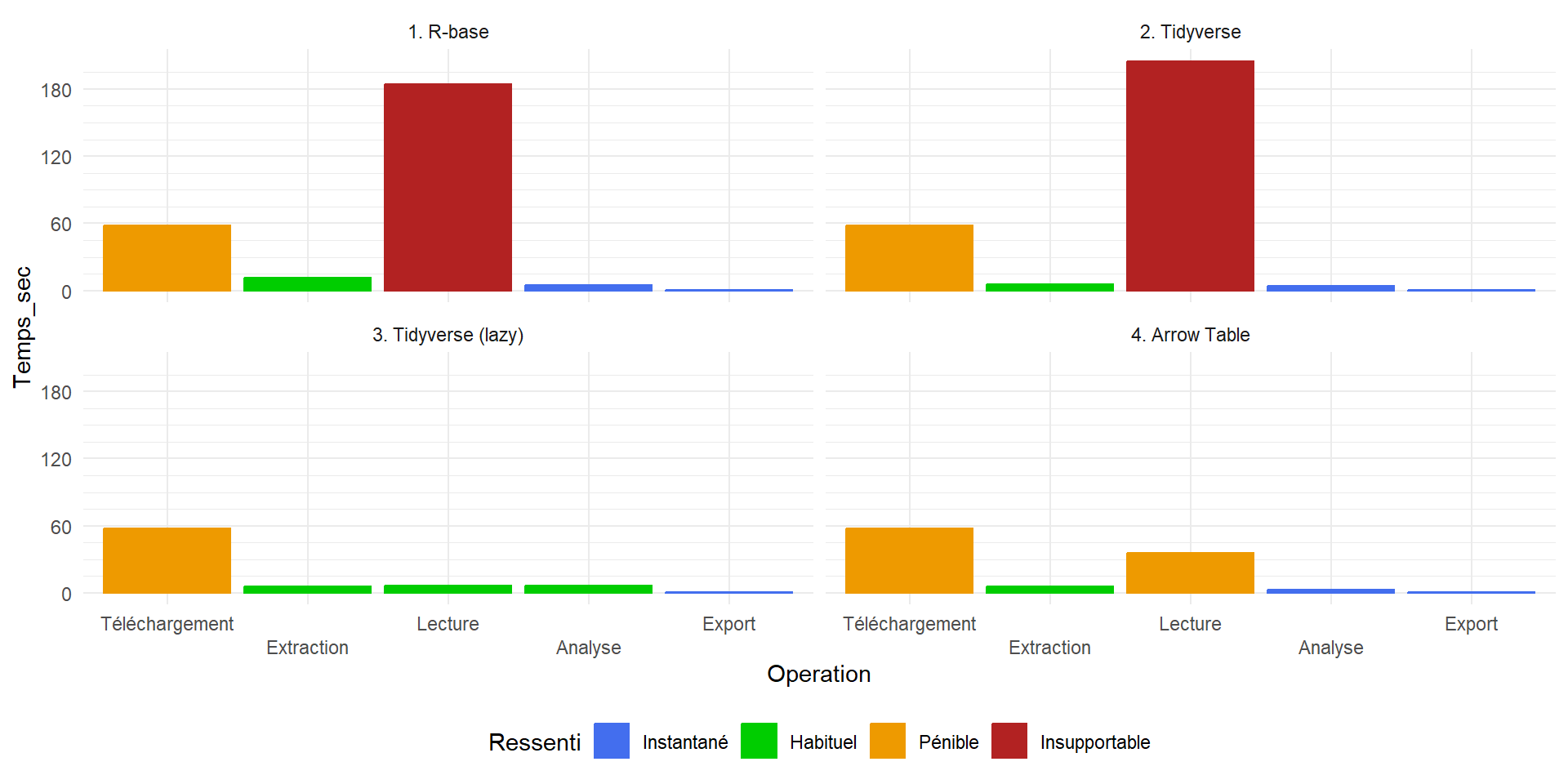
Quelle différence ?
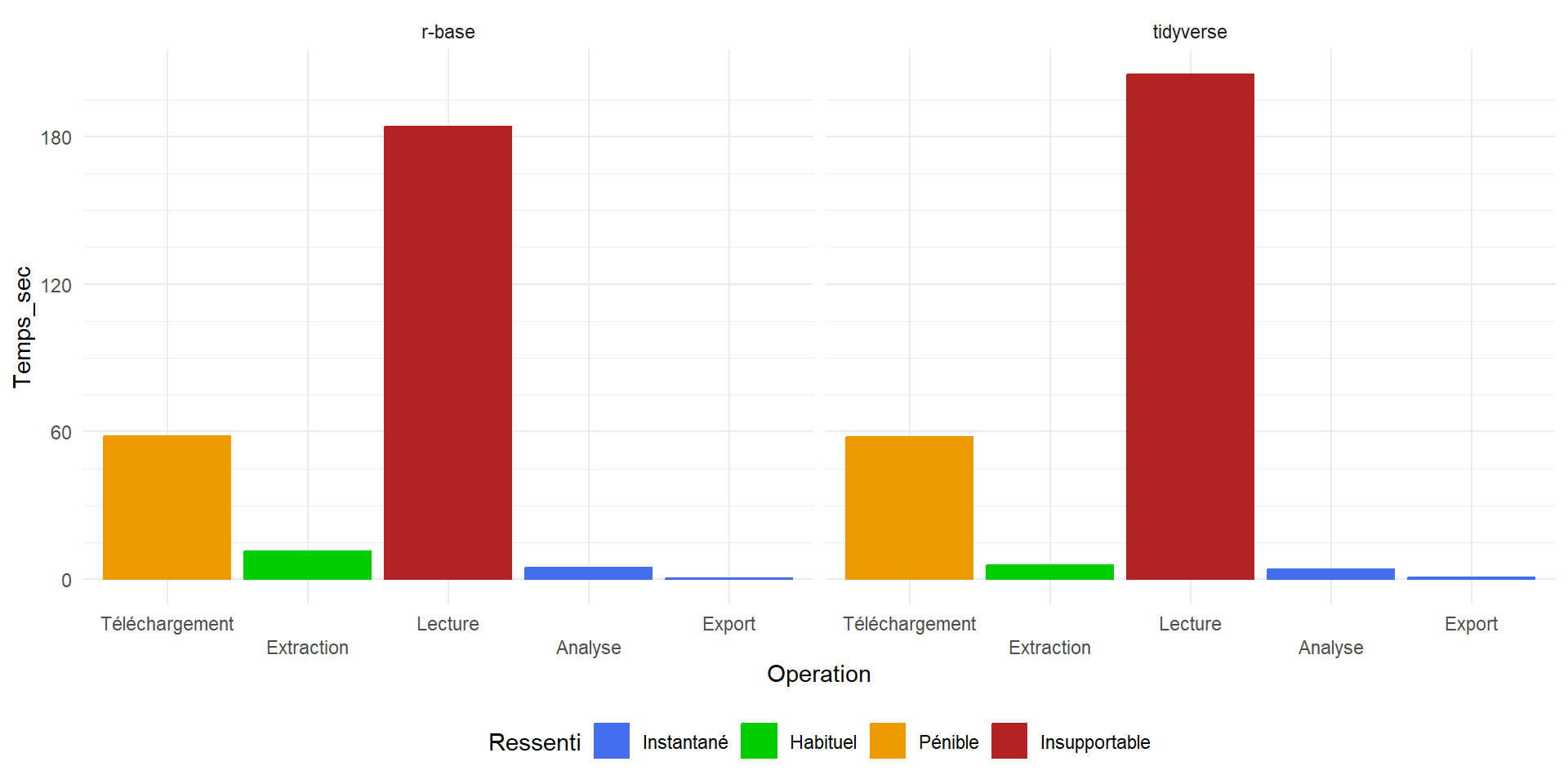
C’est quand même pas terrible !
Quel est le goulot d’étranglement (bottleneck) ?

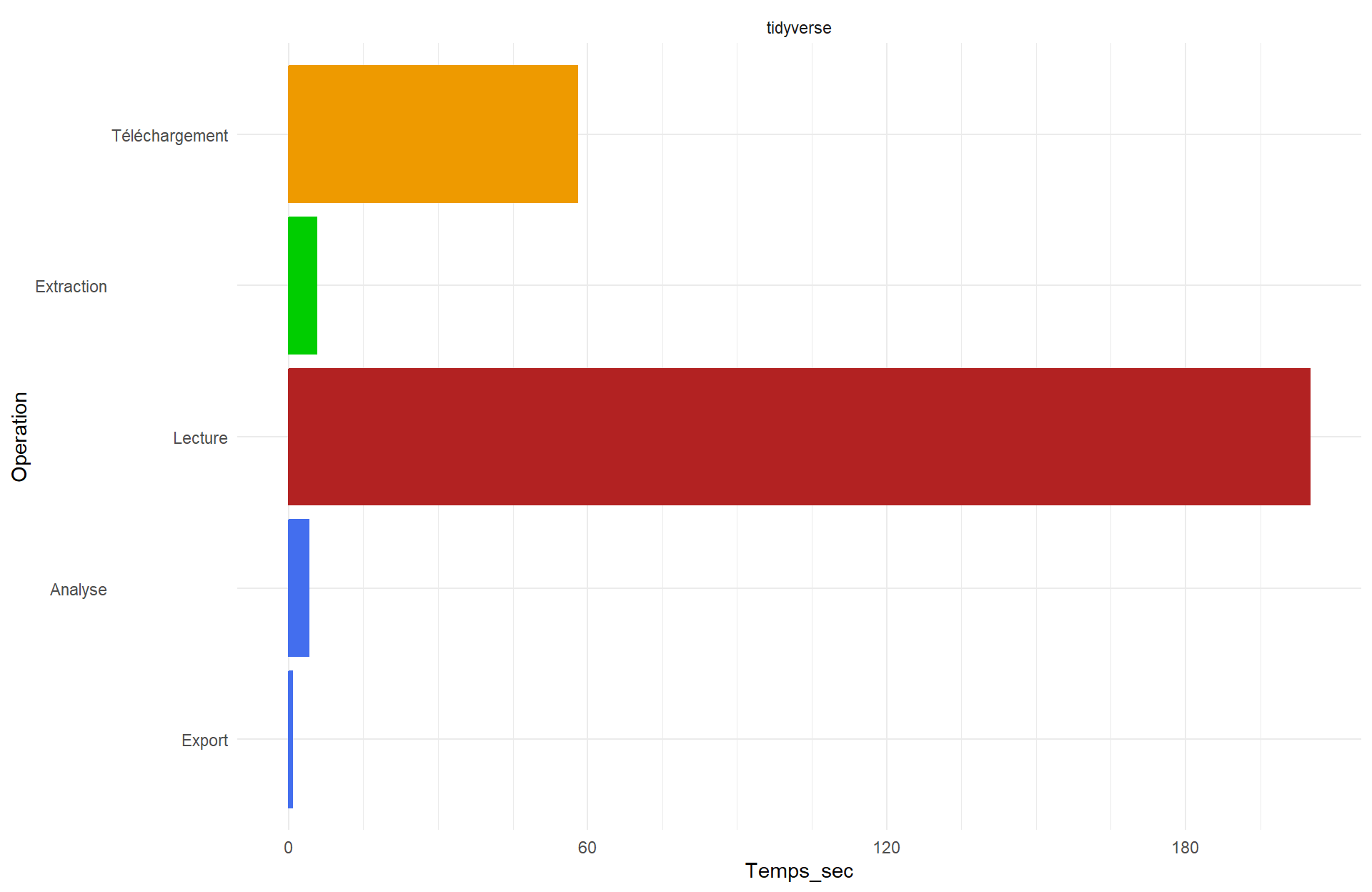
Une petite astuce
Et avec l’analyse après :
library(dplyr)
library(tidyr)
resume_commune <- rp2020 %>%
group_by(COMMUNE, CATL, TYPL) %>%
summarise(nb_logements = n(),
.groups = "drop")
resume_CATL <- resume_commune %>%
group_by(COMMUNE, CATL) %>%
summarise(nb_logements = sum(nb_logements),
.groups = "drop") %>%
pivot_wider(id_cols = COMMUNE,
names_from = CATL,names_prefix = "CATL_",
values_from = nb_logements,
values_fill = 0)resume_TYPL <- resume_commune %>%
group_by(COMMUNE, TYPL) %>%
summarise(nb_logements = sum(nb_logements),
.groups = "drop") %>%
pivot_wider(id_cols = COMMUNE,
names_from = TYPL, names_prefix = "TYPL_",
values_from = nb_logements,
values_fill = 0)
resume_global <- resume_commune %>%
group_by(COMMUNE) %>%
summarise(LOGEMENTS_TOTAL = sum(nb_logements),
.groups = "drop") %>%
left_join(resume_CATL, by = "COMMUNE") %>%
left_join(resume_TYPL, by = "COMMUNE")Statistiques
: 6.75 sec : ~13 Go en pic
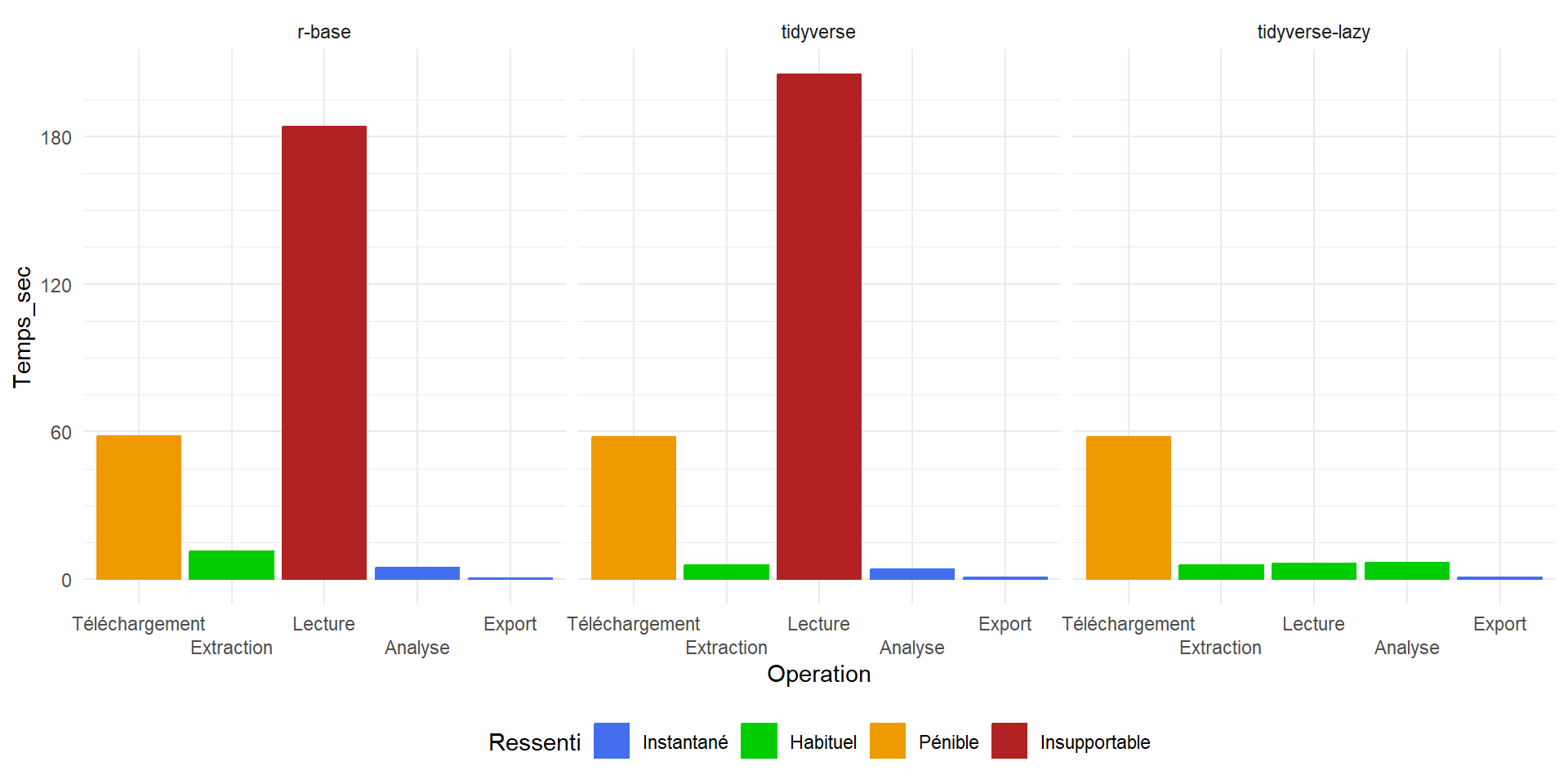
Et la mémoire vive

- R base : 25 Go en pic, 13 Go après
- Tidyverse : 22 Go en pic, 13 Go après
- Tidyverse “lazy” : 13 Go en pic, 13 Go après
- Pour toutes les méthodes présentées jusque là, ce “simple” fichier CSV de 4.5 Go occupe 13 Go de RAM
- Ça ne marchera pas sur un ordinateur ayant moins de 16 Go de RAM, et avec difficulté…
Un format de sérialisation plus performant

Apache Arrow is a language-agnostic software framework for developing data analytics applications that process columnar data. It contains a standardized column-oriented memory format that is able to represent flat and hierarchical data for efficient analytic operations on modern CPU and GPU hardware. Wikipedia, 2025

- Un format de représentation de données tabulaires en mémoire :
Arrow Table - Inter-opérable
- Orienté colonne
Retour à notre chaîne de traitement - 2. Lecture
Tidyverse
Statistiques - Lecture
: 205 sec : ~22 Go en pic, ~13 Go après
Retour à notre chaîne de traitement - 2. Lecture
Arrow
Table
25795751 rows x 69 columns
$COMMUNE <string>
$ARM <string>
$IRIS <string>
$ACHL <string>
$AEMM <int64>
$AEMMR <int64>
...
63 more columns
Use `schema()` to see entire schema[1] "Table" "ArrowTabular" "ArrowObject" "R6" - Comment vérifier le contenu de la table ?
Comment vérifier le contenu de la table ?
- Pour voir le contenu de la table, on doit utiliser la fonction
collect()qui la matérialise au format R.
# A tibble: 5 × 69
COMMUNE ARM IRIS ACHL AEMM AEMMR AGEMEN8 ANEM ANEMR ASCEN BAIN BATI CATIRIS CATL CHAU CHFL CHOS CLIM CMBL CUIS DEROU DIPLM
<chr> <chr> <chr> <chr> <int> <int> <chr> <int> <int> <chr> <chr> <chr> <chr> <int> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <chr>
1 01001 ZZZZZ ZZZZZ… A11 1993 8 65 29 4 2 Z Z Z 1 Z 2 Z Z 5 Z Z 14
2 01001 ZZZZZ ZZZZZ… C113 2013 9 40 9 2 2 Z Z Z 1 Z 3 Z Z 4 Z Z 16
3 01001 ZZZZZ ZZZZZ… C113 2013 9 65 9 2 2 Z Z Z 1 Z 4 Z Z 5 Z Z 13
4 01001 ZZZZZ ZZZZZ… A12 1994 8 65 28 4 2 Z Z Z 1 Z 2 Z Z 6 Z Z 13
5 01001 ZZZZZ ZZZZZ… C115 0 0 YY 999 99 Y Z Z Z 2 Z Y Z Z Y Z Z YY
# ℹ 47 more variables: EAU <chr>, EGOUL <chr>, ELEC <chr>, EMPLM <chr>, GARL <chr>, HLML <chr>, ILETUDM <chr>, ILTM <chr>, IMMIM <chr>,
# INAIM <chr>, INEEM <chr>, INP11M <chr>, INP15M <chr>, INP17M <chr>, INP19M <chr>, INP24M <chr>, INP3M <chr>, INP60M <chr>,
# INP65M <chr>, INP5M <chr>, INP75M <chr>, INPAM <chr>, INPER <chr>, INPER1 <chr>, INPER2 <chr>, INPOM <chr>, INPSM <chr>, IPONDL <chr>,
# IRANM <chr>, METRODOM <chr>, NBPI <chr>, RECHM <chr>, REGION <int>, SANI <chr>, SANIDOM <chr>, SEXEM <chr>, STAT_CONJM <chr>,
# STOCD <int>, SURF <chr>, TACTM <chr>, TPM <chr>, TRANSM <chr>, TRIRIS <chr>, TYPC <chr>, TYPL <int>, VOIT <chr>, WC <chr>Retour à notre chaîne de traitement - 3. Analyse
Tidyverse
library(dplyr)
library(tidyr)
resume_commune <- rp2020 %>%
group_by(COMMUNE, CATL, TYPL) %>%
summarise(nb_logements = n(),
.groups = "drop")
resume_CATL <- resume_commune %>%
group_by(COMMUNE, CATL) %>%
summarise(nb_logements = sum(nb_logements),
.groups = "drop") %>%
pivot_wider(id_cols = COMMUNE,
names_from = CATL,names_prefix = "CATL_",
values_from = nb_logements,
values_fill = 0)Arrow
library(dplyr)
library(tidyr)
resume_commune <- rp2020 %>%
group_by(COMMUNE, CATL, TYPL) %>%
summarise(nb_logements = n(),
.groups = "drop")
resume_CATL <- resume_commune %>%
group_by(COMMUNE, CATL) %>%
summarise(nb_logements = sum(nb_logements),
.groups = "drop") %>%
collect() %>%
pivot_wider(id_cols = COMMUNE,
names_from = CATL,names_prefix = "CATL_",
values_from = nb_logements,
values_fill = 0)- On peut interroger une
Arrow Tableavec les fonctionsdplyr, sans modification du code.
Retour à notre chaîne de traitement - 3. Analyse
Tidyverse
library(dplyr)
library(tidyr)
resume_commune <- rp2020 %>%
group_by(COMMUNE, CATL, TYPL) %>%
summarise(nb_logements = n(),
.groups = "drop")
resume_CATL <- resume_commune %>%
group_by(COMMUNE, CATL) %>%
summarise(nb_logements = sum(nb_logements),
.groups = "drop") %>%
pivot_wider(id_cols = COMMUNE,
names_from = CATL,names_prefix = "CATL_",
values_from = nb_logements,
values_fill = 0)Arrow
library(dplyr)
library(tidyr)
resume_commune <- rp2020 %>%
group_by(COMMUNE, CATL, TYPL) %>%
summarise(nb_logements = n(),
.groups = "drop")
resume_CATL <- resume_commune %>%
group_by(COMMUNE, CATL) %>%
summarise(nb_logements = sum(nb_logements),
.groups = "drop") %>%
collect() %>%
pivot_wider(id_cols = COMMUNE,
names_from = CATL,names_prefix = "CATL_",
values_from = nb_logements,
values_fill = 0)- On peut interroger une
Arrow Tableavec les fonctionsdplyr, presque sans modification du code.
Retour à notre chaîne de traitement - 3. Analyse
resume_commune <- rp2020 %>% group_by(COMMUNE, CATL, TYPL) %>% summarise(nb_logements = n(), .groups = "drop")
resume_CATL <- resume_commune %>%
group_by(COMMUNE, CATL) %>%
summarise(nb_logements = sum(nb_logements), .groups = "drop") %>%
collect() %>%
pivot_wider(id_cols = COMMUNE, names_from = CATL, names_prefix = "CATL_", values_from = nb_logements, values_fill = 0)
resume_TYPL <- resume_commune %>%
group_by(COMMUNE, TYPL) %>%
summarise(nb_logements = sum(nb_logements), .groups = "drop") %>%
collect() %>%
pivot_wider(id_cols = COMMUNE, names_from = TYPL, names_prefix = "TYPL_", values_from = nb_logements, values_fill = 0)
resume_global <- resume_commune %>%
group_by(COMMUNE) %>%
summarise(LOGEMENTS_TOTAL = sum(nb_logements), .groups = "drop") %>%
left_join(resume_CATL, by = "COMMUNE") %>%
left_join(resume_TYPL, by = "COMMUNE") %>%
collect()Statistiques - Analyse
: 2.67 sec : ~6 Go stable
Retour à notre chaîne de traitement - 3. Analyse
resume_commune <- rp2020 %>% group_by(COMMUNE, CATL, TYPL) %>% summarise(nb_logements = n(), .groups = "drop")
resume_CATL <- resume_commune %>%
group_by(COMMUNE, CATL) %>%
summarise(nb_logements = sum(nb_logements), .groups = "drop") %>%
collect() %>%
pivot_wider(id_cols = COMMUNE, names_from = CATL, names_prefix = "CATL_", values_from = nb_logements, values_fill = 0)
resume_TYPL <- resume_commune %>%
group_by(COMMUNE, TYPL) %>%
summarise(nb_logements = sum(nb_logements), .groups = "drop") %>%
collect() %>%
pivot_wider(id_cols = COMMUNE, names_from = TYPL, names_prefix = "TYPL_", values_from = nb_logements, values_fill = 0)
resume_global <- resume_commune %>%
group_by(COMMUNE) %>%
summarise(LOGEMENTS_TOTAL = sum(nb_logements), .groups = "drop") %>%
left_join(resume_CATL, by = "COMMUNE") %>%
left_join(resume_TYPL, by = "COMMUNE") %>%
collect()Statistiques - Analyse
: 2.67 sec : ~6 Go stable
Influence des collect() hors arrow
- Dans une chaîne de traitement
dplyrhabituelle, lecollect()n’a aucun effet et n’entraîne pas d’erreur : laisser descollect()n’entraîne aucun risque, ils sont ignorés.
Petit récapitulatif
Le temps de lecture est encore pénible.
On ne pourrait pas aussi avoir une logique d’indexation du fichier sans le charger ?
Les Dataset Arrow
Pour interroger et manipuler des fichiers sans les charger en mémoire, Arrow propose d’utiliser des Arrow Dataset à la place des Arrow Table
Table
25795751 rows x 69 columns
$COMMUNE <string>
$ARM <string>
$IRIS <string>
$ACHL <string>
$AEMM <int64>
$AEMMR <int64>
...
63 more columns
Use `schema()` to see entire schemalibrary(arrow)
csv_types <- paste0(c("c", rep("?", 68)), collapse = "")
csv_names <- readr::read_csv2("data/FD_LOGEMT_2020.csv", n_max = 2, show_col_types = FALSE) |> colnames()
rp2020_dataset <- open_dataset(
sources = "data/FD_LOGEMT_2020.csv",
delim = ";",
col_names = csv_names,
col_types = csv_types
)
rp2020_datasetFileSystemDataset with 1 csv file
69 columns
COMMUNE: string
ARM: string
IRIS: string
ACHL: string
AEMM: string
AEMMR: string
...
63 more columns
Use `schema()` to see entire schemaLes Dataset Arrow
Pour interroger et manipuler des fichiers sans les charger en mémoire, Arrow propose d’utiliser des Arrow Dataset à la place des Arrow Table
Statistiques - Lecture
: 36 sec : ~18 Go en pic, ~6 Go après
. . .
Et pour le traitement, comme pour des Table Arrow, on récupère les données avec un collect().
resume_commune <- rp2020_dataset %>%
group_by(COMMUNE, CATL, TYPL) %>%
summarise(nb_logements = n(), .groups = "drop")
resume_CATL <- resume_commune %>%
group_by(COMMUNE, CATL) %>%
summarise(nb_logements = sum(nb_logements), .groups = "drop") %>%
collect() %>%
pivot_wider(id_cols = COMMUNE, names_from = CATL, names_prefix = "CATL_", values_from = nb_logements, values_fill = 0)
resume_TYPL <- resume_commune %>%
group_by(COMMUNE, TYPL) %>%
summarise(nb_logements = sum(nb_logements), .groups = "drop") %>%
collect() %>%
pivot_wider(id_cols = COMMUNE, names_from = TYPL, names_prefix = "TYPL_", values_from = nb_logements, values_fill = 0)
resume_global <- resume_commune %>%
group_by(COMMUNE) %>%
summarise(LOGEMENTS_TOTAL = sum(nb_logements), .groups = "drop") %>%
left_join(resume_CATL, by = "COMMUNE") %>%
left_join(resume_TYPL, by = "COMMUNE") %>%
collect()Statistiques - Lecture
: 17.00 sec : 750 Mo en pic
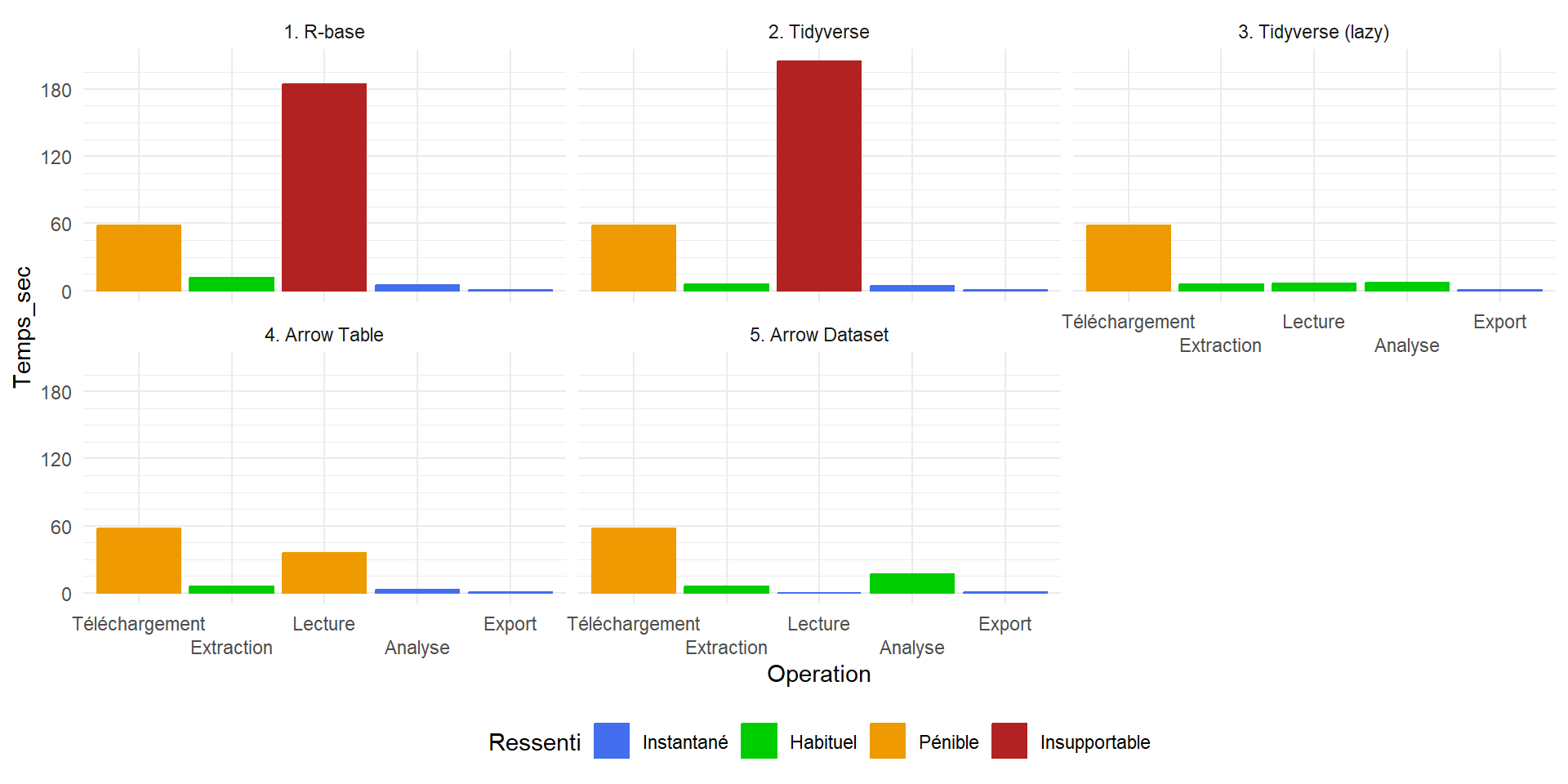
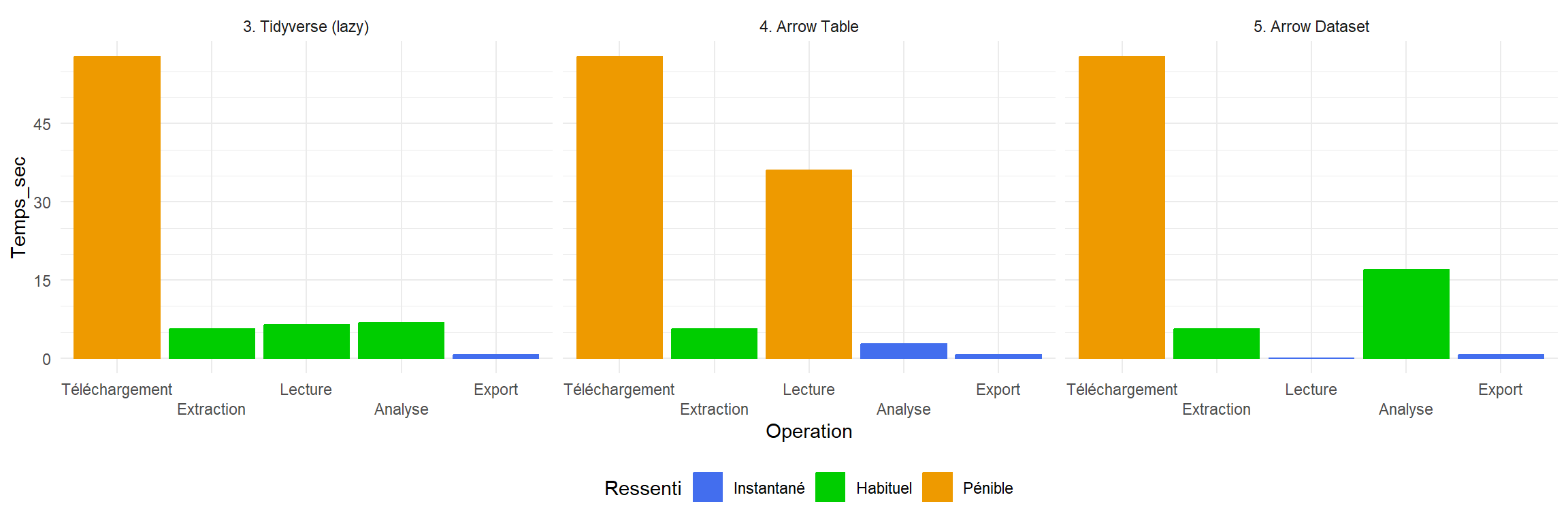
Lecture + analyse :
Tidyverse lazy
: 12 sec : 13 Go (max)
Arrow Table
: 42 sec : 6 Go (max)
Arrow Dataset (lazy)
: 6 sec : 750 Mo (max)
Le problème avec arrow
library(arrow)
csv_types <- paste0(c("c", rep("?", 68)), collapse = "")
csv_names <- readr::read_csv2("data/FD_LOGEMT_2020.csv", n_max = 2, show_col_types = FALSE) |> colnames()
rp2020_dataset <- open_dataset(
sources = "data/FD_LOGEMT_2020.csv",
delim = ";",
col_names = csv_names, col_types = csv_types)
rp2020_dataset %>%
group_by(COMMUNE) %>%
slice_max(NBPI, n = 1)Error in `arrow_not_supported()`:
! Slicing grouped data not supported in ArrowLe problème avec arrow
Error in `arrow_not_supported()`:
! Slicing grouped data not supported in ArrowArrow permet de stocker des tableaux en mémoire et des les interroger avec des fonctionnalités spécifiques.
Toutes les fonctions de
dplyrne sont pas intégrées dans arrow, notamment les fonctions “fenêtrées” (window function).Arrow est un formidable outil pour manipuler/agréger des données (très) massives avec une empreinte mémoire extrêmement réduite, mais c’est avant tout un format de données en mémoire vive, qui sert de base à de nombreux autres outils.
Un outil versatile et performant d’analyse de données massives : DuckDB

DuckDB is an open-source column-oriented Relational Database Management System (RDBMS). It is designed to provide high performance on complex queries against large databases in embedded configuration, such as combining tables with hundreds of columns and billions of rows. Wikipedia, 2025
- Un Système de Gestion de Base de Données (SGBD) relationnel
- Orienté colonne
- Portable
- Permettant une interrogation structurée (SQL)
Utilisation de DuckDB
- DuckDB est un système de base de données qu’il faut initialiser en démarrant une connexion
Utilisation de DuckDB
- DuckDB est un système de base de données qu’il faut initialiser en démarrant une connexion
- Et auquel on peut envoyer des instructions SQL avec
dbSendQuery().
Statistiques - Lecture
: 25.56 sec : 15 Go
Utilisation de DuckDB en SQL
- Avec ce format natif, on va se contenter d’utiliser R pour exécuter des requêtes SQL :
dbSendQuery(con,
"CREATE TABLE resume_commune AS
SELECT COMMUNE, CATL, TYPL, COUNT(*) as nb_logements
FROM rp2020
GROUP BY COMMUNE, CATL, TYPL;"
)
dbSendQuery(con,
"CREATE TABLE resume_CATL AS
SELECT COMMUNE, CATL, SUM(nb_logements) as nb_logements
FROM resume_commune
GROUP BY COMMUNE, CATL;"
)
dbSendQuery(con,
"CREATE TABLE resume_TYPL AS
SELECT COMMUNE, TYPL, SUM(nb_logements) as nb_logements
FROM resume_commune
GROUP BY COMMUNE, TYPL;"
)
dbSendQuery(con,
"CREATE TABLE resume_CATL_wide AS
SELECT
COMMUNE,
MAX(CASE WHEN (CATL = 1.0) THEN nb_logements WHEN NOT (CATL = 1.0) THEN 0.0 END) AS CATL_1,
MAX(CASE WHEN (CATL = 2.0) THEN nb_logements WHEN NOT (CATL = 2.0) THEN 0.0 END) AS CATL_2,
MAX(CASE WHEN (CATL = 3.0) THEN nb_logements WHEN NOT (CATL = 3.0) THEN 0.0 END) AS CATL_3,
MAX(CASE WHEN (CATL = 4.0) THEN nb_logements WHEN NOT (CATL = 4.0) THEN 0.0 END) AS CATL_4
FROM resume_CATL
GROUP BY COMMUNE;"
)dbSendQuery(con,
"CREATE TABLE resume_TYPL_wide AS
SELECT
COMMUNE,
MAX(CASE WHEN (TYPL = 1.0) THEN nb_logements WHEN NOT (TYPL = 1.0) THEN 0.0 END) AS TYPL_1,
MAX(CASE WHEN (TYPL = 2.0) THEN nb_logements WHEN NOT (TYPL = 2.0) THEN 0.0 END) AS TYPL_2,
MAX(CASE WHEN (TYPL = 3.0) THEN nb_logements WHEN NOT (TYPL = 3.0) THEN 0.0 END) AS TYPL_3,
MAX(CASE WHEN (TYPL = 4.0) THEN nb_logements WHEN NOT (TYPL = 4.0) THEN 0.0 END) AS TYPL_4,
MAX(CASE WHEN (TYPL = 5.0) THEN nb_logements WHEN NOT (TYPL = 5.0) THEN 0.0 END) AS TYPL_5,
MAX(CASE WHEN (TYPL = 6.0) THEN nb_logements WHEN NOT (TYPL = 6.0) THEN 0.0 END) AS TYPL_6
FROM resume_TYPL
GROUP BY COMMUNE"
)
dbSendQuery(con,
"CREATE TABLE resume_commune_2 AS
SELECT COMMUNE, SUM(nb_logements) AS LOGEMENTS_TOTAL
FROM resume_commune
GROUP BY COMMUNE;"
)
dbSendQuery(con,
"CREATE TABLE resume_global AS
SELECT res2.COMMUNE, res2.LOGEMENTS_TOTAL,
CATL_1, CATL_2, CATL_3, CATL_4,
TYPL_1, TYPL_2, TYPL_3, TYPL_4, TYPL_5, TYPL_6
FROM resume_commune_2 res2
LEFT JOIN resume_CATL_wide catl
ON res2.COMMUNE = catl.COMMUNE
LEFT JOIN resume_TYPL_wide typl
ON res2.COMMUNE = typl.COMMUNE;"
)Utilisation de DuckDB en SQL
- Comme pour arrow, on ne récupère le résultat final en “format R” qu’à la fin de notre chaîne de traitement, avec
dbGetQuery
'data.frame': 34933 obs. of 12 variables:
$ COMMUNE : chr "01001" "01002" "01004" "01005" ...
$ LOGEMENTS_TOTAL: int 379 175 3059 832 71 1261 354 191 591 187 ...
$ CATL_1 : int 354 121 2753 736 57 1169 322 145 464 150 ...
$ CATL_2 : int 1 0 30 0 0 5 0 1 2 1 ...
$ CATL_3 : int 10 41 15 7 8 36 6 23 76 20 ...
$ CATL_4 : int 14 13 261 89 6 51 26 22 49 16 ...
$ TYPL_1 : int 369 173 1098 614 68 1070 323 178 530 163 ...
$ TYPL_2 : int 9 2 1938 215 3 189 31 12 59 23 ...
$ TYPL_6 : int 1 0 4 0 0 1 0 0 1 0 ...
$ TYPL_3 : int 0 0 17 1 0 0 0 0 1 0 ...
$ TYPL_5 : int 0 0 2 2 0 1 0 1 0 1 ...
$ TYPL_4 : int 0 0 0 0 0 0 0 0 0 0 ...Statistiques - Analyse
: 0.73 sec : 15 Go
Utilisation de DuckDB avec dplyr
Le SQL est un langage de requête universel et standardisé.
C’est un langage de manipulation de données, et pas de programmation : la syntaxe de
dplyren est très inspiréeTellement inspirée qu’avec
dplyr, on peut même exprimer des requêtes en SQL :
<SQL>
SELECT cyl, AVG(mpg) AS mean_mpg, MEDIAN(mpg) AS med_mpg
FROM mtcars
GROUP BY cylUtilisation de DuckDB avec dplyr
Le SQL est un langage de requête universel et standardisé.
C’est un langage de manipulation de données, et pas de programmation : la syntaxe de
dplyren est très inspiréeTellement inspirée qu’avec
dplyr, on peut même exprimer des requêtes en SQL.
Tip
C’est avec cette capacité de dplyr à convertir ses instructions en SQL qu’on a pu utiliser
arrowavec une syntaxe dplyr…Et qu’on peut l’utiliser avec
DuckDB!
Utilisation de DuckDB avec dplyr
- Lecture
# Source: SQL [?? x 69]
# Database: DuckDB v1.2.1 [robin@Windows 10 x64:R 4.4.1/:memory:]
COMMUNE ARM IRIS ACHL AEMM AEMMR AGEMEN8 ANEM ANEMR ASCEN BAIN BATI CATIRIS CATL CHAU CHFL CHOS
<chr> <chr> <chr> <chr> <chr> <dbl> <chr> <chr> <chr> <chr> <chr> <chr> <chr> <dbl> <chr> <chr> <chr>
1 01001 ZZZZZ ZZZZ… A11 1993 8 65 029 04 2 Z Z Z 1 Z 2 Z
2 01001 ZZZZZ ZZZZ… C113 2013 9 40 009 02 2 Z Z Z 1 Z 3 Z
3 01001 ZZZZZ ZZZZ… C113 2013 9 65 009 02 2 Z Z Z 1 Z 4 Z
4 01001 ZZZZZ ZZZZ… A12 1994 8 65 028 04 2 Z Z Z 1 Z 2 Z
5 01001 ZZZZZ ZZZZ… C115 0000 0 YY 999 99 Y Z Z Z 2 Z Y Z
6 01001 ZZZZZ ZZZZ… A12 2021 9 40 001 00 2 Z Z Z 1 Z 2 Z
7 01001 ZZZZZ ZZZZ… A12 2021 9 65 001 00 2 Z Z Z 1 Z 2 Z
8 01001 ZZZZZ ZZZZ… C100 2018 9 40 004 01 2 Z Z Z 1 Z 2 Z
9 01001 ZZZZZ ZZZZ… A11 2020 9 25 002 01 2 Z Z Z 1 Z 2 Z
10 01001 ZZZZZ ZZZZ… A11 1986 7 65 036 05 2 Z Z Z 1 Z 4 Z
# ℹ more rows
# ℹ 52 more variables: CLIM <chr>, CMBL <chr>, CUIS <chr>, DEROU <chr>, DIPLM <chr>, EAU <chr>, EGOUL <chr>,
# ELEC <chr>, EMPLM <chr>, GARL <chr>, HLML <chr>, ILETUDM <chr>, ILTM <chr>, IMMIM <chr>, INAIM <chr>,
# INEEM <chr>, INP11M <chr>, INP15M <chr>, INP17M <chr>, INP19M <chr>, INP24M <chr>, INP3M <chr>,
# INP60M <chr>, INP65M <chr>, INP5M <chr>, INP75M <chr>, INPAM <chr>, INPER <chr>, INPER1 <chr>,
# INPER2 <chr>, INPOM <chr>, INPSM <chr>, IPONDL <dbl>, IRANM <chr>, METRODOM <chr>, NBPI <chr>,
# RECHM <chr>, REGION <dbl>, SANI <chr>, SANIDOM <chr>, SEXEM <chr>, STAT_CONJM <chr>, STOCD <chr>, …Utilisation de DuckDB avec dplyr
- Analyse
resume_commune <- rp2020 %>%
group_by(COMMUNE, CATL, TYPL) %>%
summarise(nb_logements = n(), .groups = "drop")
resume_CATL <- resume_commune %>%
group_by(COMMUNE, CATL) %>%
summarise(nb_logements = sum(nb_logements), .groups = "drop") %>%
pivot_wider(id_cols = COMMUNE, names_from = CATL, names_prefix = "CATL_", values_from = nb_logements, values_fill = 0)
resume_TYPL <- resume_commune %>%
group_by(COMMUNE, TYPL) %>%
summarise(nb_logements = sum(nb_logements), .groups = "drop") %>%
pivot_wider(id_cols = COMMUNE, names_from = TYPL, names_prefix = "TYPL_", values_from = nb_logements, values_fill = 0)
resume_global <- resume_commune %>%
group_by(COMMUNE) %>%
summarise(LOGEMENTS_TOTAL = sum(nb_logements), .groups = "drop") %>%
left_join(resume_CATL, by = "COMMUNE") %>%
left_join(resume_TYPL, by = "COMMUNE") %>%
collect()Utilisation de DuckDB avec dplyr
Statistiques - Lecture
: 25.56 sec : 15 Go
Statistiques - Analyse
: 2.75 sec : 15 Go
Utilisation de DuckDB avec dplyr en indexation
- Lecture/indexation
Statistiques - Indexation
: 0.31 sec : 0 Mo
- Analyse (idem précédent)
Statistiques - Indexation
: 12.44 sec : ~200 Mo
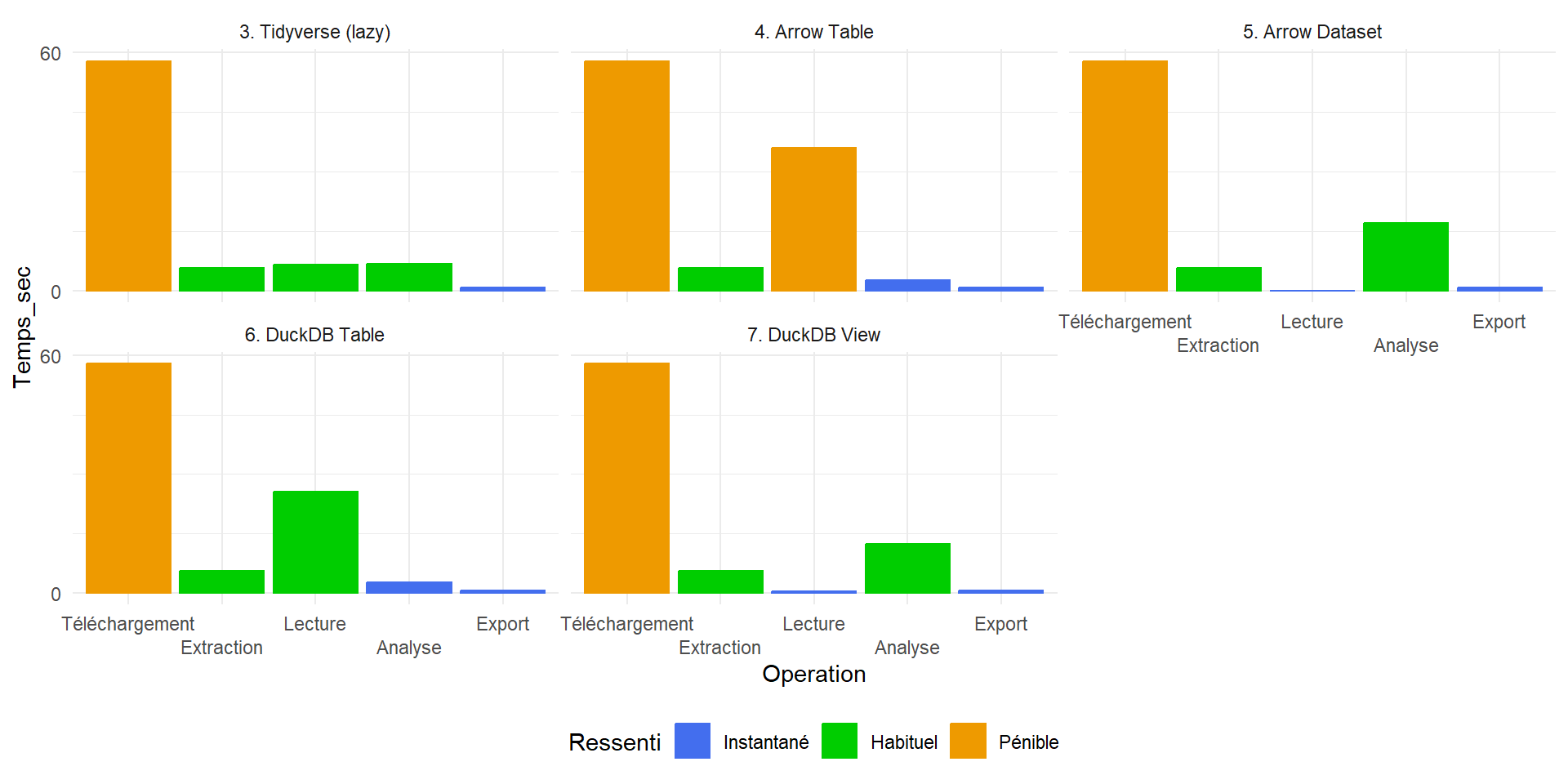
Encore un récapitulatif
Point d’étape
Arrowest un format de données en mémoire, qui permet d’interroger des données massives rapidement et avec une empreinte mémoire très réduite mais des capacités inférieures à dplyr.DuckDBest un système de gestion de bases de données en mémoire et propose ~ toutes les opérations dedplyravec une empreinte mémoire très réduite et un temps de traitement très rapide.
Point d’étape
Avec ces deux systèmes concurrents*, on arrive à traiter très rapidement des données tabulaires importantes, avec deux voies alternatives :
Lire entièrement le fichier en entrée (36 sec Arrow, 25 DuckDB) et l’analyser depuis la table créée (2.67 sec Arrow, 2.75 DuckDB)
Indexer le fichier (quasi instantanée) et l’analyser ensuite depuis le dataset/view générées (17 sec Arrow, 12 DuckDB)
Le choix de la méthode dépend du traitement voulu et de la taille des données :
- si on doit faire de nombreux traitements successifs sur un même jeu de données et qu’il tient en mémoire vive, on le lit.
- Sinon, on l’indexe.
Comment gagner encore du temps et des capacités à traiter de plus gros jeux de données ?
Réduire la taille des fichiers manipulés pour ne pas avoir à archiver/désarchiver les données
Accélérer la lecture des fichiers pour les traiter en mémoire
Accélérer le traitement de fichiers indexés
Se passer des phases de téléchargement
Comment gagner encore du temps et des capacités à traiter de plus gros jeux de données ?
Réduire la taille des fichiers manipulés pour ne pas avoir à archiver/désarchiver les données
Accélérer la lecture des fichiers pour les traiter en mémoire
Accélérer le traitement de fichiers indexés
Se passer des phases de téléchargement
Pour tout ça, il y a le format Parquet !
Apache Parquet
Apache Parquet est un format de fichiers orienté colonne, initialement développé pour l’écosystème de calcul distribué Apache Hadoop. […] Il fournit des schémas efficaces de compression et de codage de données avec des performances améliorées pour gérer des données complexes en masse.
Wikipedia, 2025
- Un format de données compressé, performant, et orienté colonne.
Apache Parquet
- Un format de données compressé, performant, et orienté colonne.
Pour rappel :


Orienté colonne ?
Fichier CSV
row_names,mpg,cyl,disp,hp,drat,wt,qsec,vs,am,gear,carb
Mazda RX4,21,6,160,110,3.9,2.62,16.46,0,1,4,4
Mazda RX4 Wag,21,6,160,110,3.9,2.875,17.02,0,1,4,4
Datsun 710,22.8,4,108,93,3.85,2.32,18.61,1,1,4,1
Hornet 4 Drive,21.4,6,258,110,3.08,3.215,19.44,1,0,3,1
Hornet Sportabout,18.7,8,360,175,3.15,3.44,17.02,0,0,3,2Extrait SQL
INSERT INTO `mtcars`
(`row_names`, `mpg`, `cyl`, `disp`, `hp`, `drat`, `wt`, `qsec`, `vs`, `am`, `gear`, `carb`)
VALUES
('Mazda RX4', 21, 6, 160, 110, 3.9, 2.62, 16.46, 0, 1, 4, 4),
('Mazda RX4 Wag', 21, 6, 160, 110, 3.9, 2.875, 17.02, 0, 1, 4, 4),
('Datsun 710', 22.8, 4, 108, 93, 3.85, 2.32, 18.61, 1, 1, 4, 1),
('Hornet 4 Drive', 21.4, 6, 258, 110, 3.08, 3.215, 19.44, 1, 0, 3, 1),
('Hornet Sportabout', 18.7, 8, 360, 175, 3.15, 3.44, 17.02, 0, 0, 3, 2),Fichier JSON
[
{
"am": 1,
"carb": 4,
"cyl": 6,
"disp": 160,
"drat": 3.9,
"gear": 4,
"hp": 110,
"mpg": 21,
"qsec": 16.46,
"row_name": "Mazda RX4",
"vs": 0,
"wt": 2.62
},
{
"am": 1,
"carb": 4,
"cyl": 6,
"disp": 160,
"drat": 3.9,
"gear": 4,
"hp": 110,
"mpg": 21,
"qsec": 17.02,
"row_name": "Mazda RX4 Wag",
"vs": 0,
"wt": 2.875
},
{
"am": 1,
"carb": 1,
"cyl": 4,
"disp": 108,
"drat": 3.85,
"gear": 4,
"hp": 93,
"mpg": 22.8,
"qsec": 18.61,
"row_name": "Datsun 710",
"vs": 1,
"wt": 2.32
},
{
"am": 0,
"carb": 1,
"cyl": 6,
"disp": 258,
"drat": 3.08,
"gear": 3,
"hp": 110,
"mpg": 21.4,
"qsec": 19.44,
"row_name": "Hornet 4 Drive",
"vs": 1,
"wt": 3.215
},
{
"am": 0,
"carb": 2,
"cyl": 8,
"disp": 360,
"drat": 3.15,
"gear": 3,
"hp": 175,
"mpg": 18.7,
"qsec": 17.02,
"row_name": "Hornet Sportabout",
"vs": 0,
"wt": 3.44
}
]Orienté colonne ?
Dans un fichier classique, l’information est organisée ligne par ligne, ce qui en rend simple l’ajout ou la suppression.
C’est le modèle utilisé dans les bases de données relationnelles et pour les fichiers qui seront amenés à être modifiés fréquemment : le plus important dans ce cas est de rapidement pouvoir ajouter des lignes, ou modifier une valeur spécifique d’une ligne définie.
Cela implique aussi que pour extraire une seule colonne d’un tableau, il faut lire ce tableau en entier puis reconstituer la colonne à partir de toutes les valeurs comprises dans les lignes.
De même si on veut ajouter une colonne dans un fichier : il faut lire toutes les lignes et ajouter la valeur à la fin de chacune.
Orienté colonne ?
Pour un usage “analytique”, on utilise plus souvent une organisation orientée colonne
Voir par exemple la manière dont un
data.frameest organisé en R :
structure(list(mpg = c(21, 21, 22.8, 21.4, 18.7, 18.1), cyl = c(6,
6, 4, 6, 8, 6), disp = c(160, 160, 108, 258, 360, 225), hp = c(110,
110, 93, 110, 175, 105), drat = c(3.9, 3.9, 3.85, 3.08, 3.15,
2.76), wt = c(2.62, 2.875, 2.32, 3.215, 3.44, 3.46), qsec = c(16.46,
17.02, 18.61, 19.44, 17.02, 20.22), vs = c(0, 0, 1, 1, 0, 1),
am = c(1, 1, 1, 0, 0, 0), gear = c(4, 4, 4, 3, 3, 3), carb = c(4,
4, 1, 1, 2, 1)), row.names = c("Mazda RX4", "Mazda RX4 Wag",
"Datsun 710", "Hornet 4 Drive", "Hornet Sportabout", "Valiant"
), class = "data.frame")Orienté colonne ?
Que ce soit pour l’organisation d’un tableau en mémoire ou dans sa matérialisation en fichier, un jeu de données orienté colonne permet donc une interrogation plus rapide de chaque colonne, mais aussi :
- De ne pas lire tout un jeu de données pour extraire une seule colonne
- C’est pour cela (entre autre) que
DuckDBetArrowtraitent plus rapidement l’information
- C’est pour cela (entre autre) que
Orienté colonne ?
Que ce soit pour l’organisation d’un tableau en mémoire ou dans sa matérialisation en fichier, un jeu de données orienté colonne permet donc une interrogation plus rapide de chaque colonne, mais aussi :
- De faciliter la compression des informations
- Les fichiers orientés colonnes, quand le nombre de lignes est important, sont toujours plus légers que leurs homologues orientés lignes.
Exemple sur la couche des bâtiments de la BD TOPO :
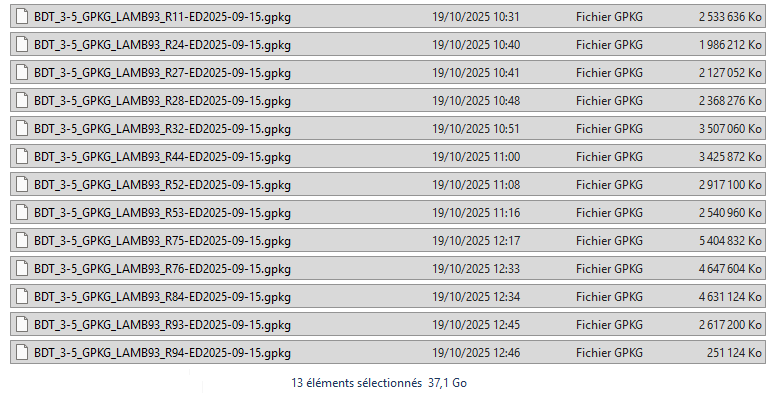
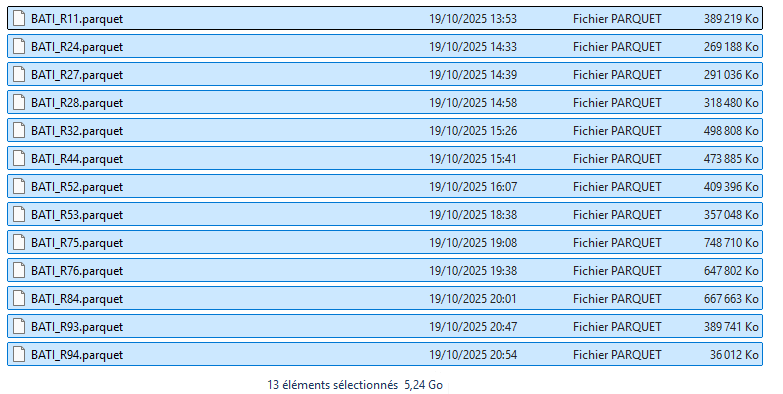
Revenons à notre exemple de chaîne de traitement
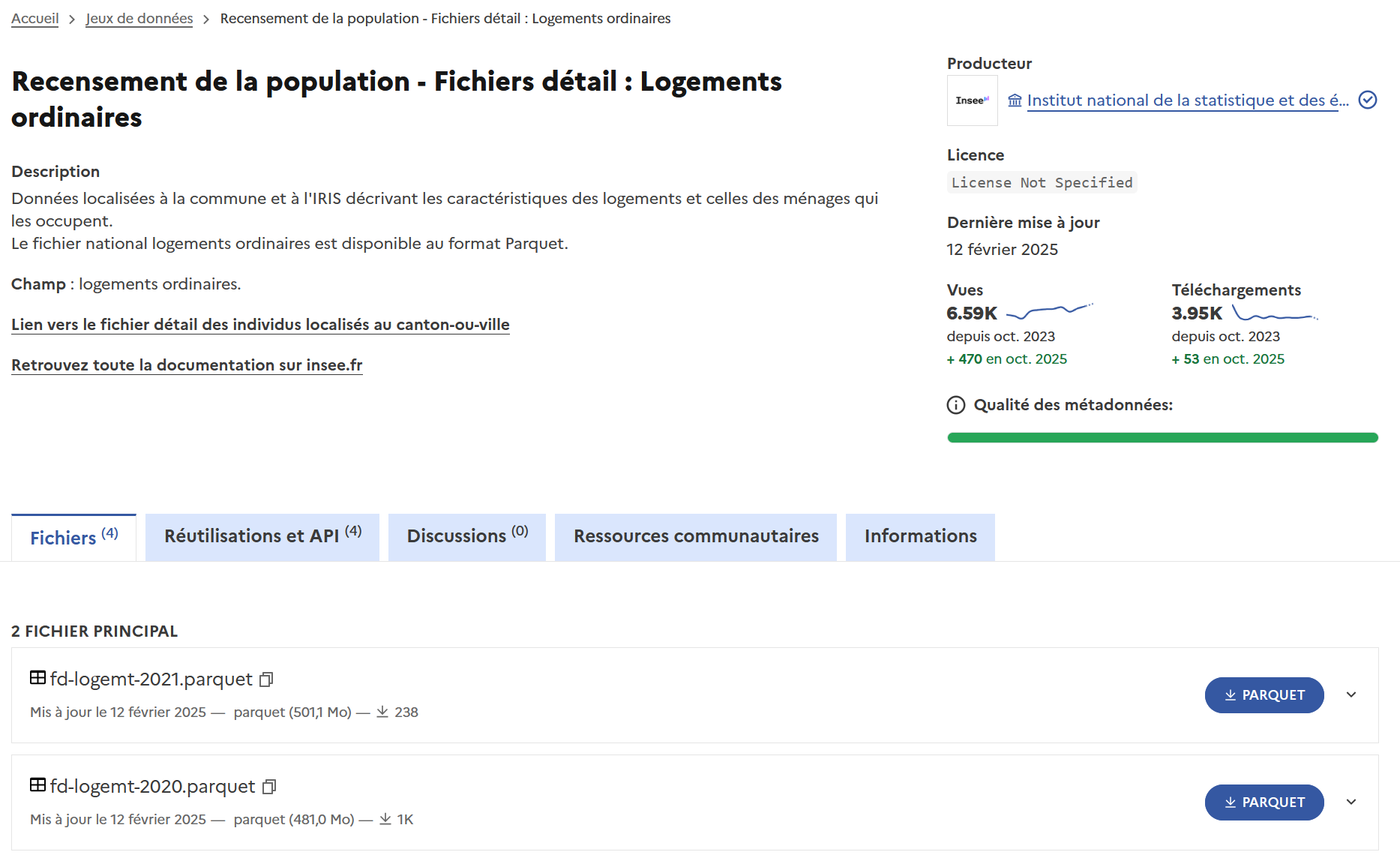
- L’INSEE propose désormais, pour certains jeux de données récents, un export au format Parquet :
Revenons à notre exemple de chaîne de traitement
- Téléchargement
Statistiques
: 25.33 sec (le serveur data.gouv est beaucoup plus rapide que celui de l’insee) : 481 Mo (un peu plus gros que le CSV.zip de 372 Mo)
Revenons à notre exemple de chaîne de traitement
Extraction : Pas nécessaire !
Lecture avec
DuckDB
Statistiques
: 6.53 sec : 14 Go
- Analyse (aucun changement dans le code)
Statistiques
: 1.45 sec : 14 Go
Et en indexant le fichier parquet ?
- Lecture avec
DuckDB
Statistiques
: 0.1 sec : ~50 Mo
- Analyse (aucun changement dans le code)
Statistiques
: 1.17 sec : ~450 Mo
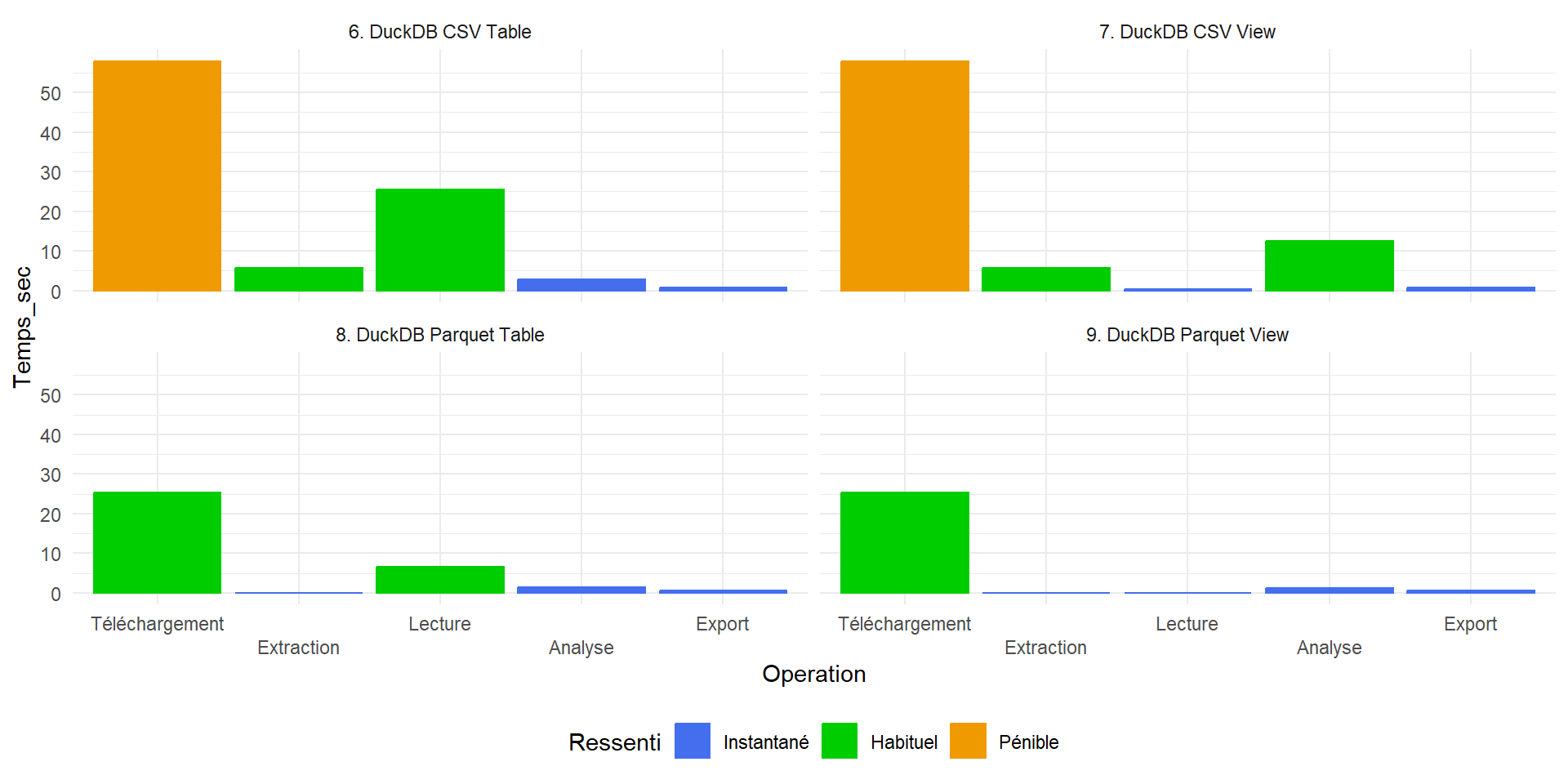
Benchmark
Une dernière chose
- Les fichiers parquet sont organisés en colonne et disposent de métadonnées extrêmement efficaces qui les rendent utilisable en streaming : pour interroger une seule colonne, on n’a pas besoin de lire tout le fichier, et on n’a pas besoin non plus de le télécharger dans son entièreté
Une dernière chose
- Pour interroger un fichier en ligne sans le télécharger,
DuckDBpropose une extensionhttpfs:
library(duckdb)
library(glue)
con <- dbConnect(duckdb())
dbSendQuery(con, "INSTALL httpfs;")
dbSendQuery(con, "LOAD httpfs;")
url_parquet <- "https://static.data.gouv.fr/resources/recensement-de-la-population-fichiers-detail-logements-ordinaires-en-2020-1/20231023-123618/fd-logemt-2020.parquet"
dbSendQuery(con, glue("CREATE OR REPLACE VIEW rp2020 AS SELECT * FROM read_parquet('{url_parquet}');"))
rp2020 <- tbl(con, "rp2020")Une dernière chose
0-3. Chaîne de traitement complète
Statistiques
: 0.22 sec (connexion) + 1.25 sec (analyse) : ~300 Mo en pic : ~230 Ko pour l’indexation + 9 Mo pour l’analyse
Une dernière chose
Au lieu de télécharger les 481 Mo du fichier Parquet en ligne, on lui a demandé uniquement ce qui était nécessaire pour l’analyse à mener : 9 Mo
Temps initial (R base) d’analyse complète : 258 secondes
Temps final avec un fichier Parquet indexé et interrogé via DuckDB : 1.47 secondes
Au delà de la rapidité, DuckDB et Arrow permettent surtout de travailler sur des jeux de données bien plus importants que la mémoire vive de votre ordinateur ne devrait le permettre
Un exemple d’analyse sur un jeu de données très massif
library(tidyverse)
library(duckdb)
# Chargement de l'extension httpfs pour lire des fichiers parquets distants
con <- dbConnect(duckdb())
dbSendQuery(con, "INSTALL httpfs;")
dbSendQuery(con, "LOAD httpfs;")
# Récupération des liens vers les fichiers parquet distant : couche des bâtiments de la BD TOPO
library(rvest)
bati_files <- read_html("https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/") %>%
html_elements("a") %>%
html_attr("href")
# Construction de la syntaxe attendue : ['https://url1', 'https://url2 etc.']
files_array <- bati_files %>% paste0("'https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/", ., "'") %>% paste0(collapse = ",\n") %>% paste0('[', ., ']')
cat(files_array)['https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/BATI_R11.parquet',
'https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/BATI_R24.parquet',
'https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/BATI_R27.parquet',
'https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/BATI_R28.parquet',
'https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/BATI_R32.parquet',
'https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/BATI_R44.parquet',
'https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/BATI_R52.parquet',
'https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/BATI_R53.parquet',
'https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/BATI_R75.parquet',
'https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/BATI_R76.parquet',
'https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/BATI_R84.parquet',
'https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/BATI_R93.parquet',
'https://analytics.huma-num.fr/Robin.Cura/ElementR_DonneesMassives/BATI_BDTOPO/BATI_R94.parquet']Un exemple d’analyse sur un jeu de données très massif
# Source: table<bati> [?? x 31]
# Database: DuckDB v1.3.0 [robin@Windows 10 x64:R 4.5.1/:memory:]
cleabs nature usage_1 usage_2 construction_legere etat_de_l_objet
<chr> <chr> <chr> <chr> <lgl> <chr>
1 BATIMENT000000033… Indif… Sportif <NA> FALSE En service
2 BATIMENT000000033… Indif… Sportif Commer… FALSE En service
3 BATIMENT000000033… Indif… Sportif Commer… FALSE En service
4 BATIMENT000000033… Indif… Sportif Réside… FALSE En service
5 BATIMENT000000033… Indif… Sportif <NA> FALSE En service
6 BATIMENT000000033… Indif… Sportif <NA> FALSE En service
# ℹ more rows
# ℹ 25 more variables: date_creation <dttm>, date_modification <dttm>,
# date_d_apparition <date>, date_de_confirmation <date>, sources <chr>,
# identifiants_sources <chr>, methode_d_acquisition_planimetrique <chr>,
# methode_d_acquisition_altimetrique <chr>, precision_planimetrique <dbl>,
# precision_altimetrique <dbl>, nombre_de_logements <int>,
# nombre_d_etages <int>, materiaux_des_murs <chr>, …Un exemple d’analyse sur un jeu de données très massif
# A tibble: 96 × 4
CODE_DEP NOM_DEP nb_batiments hauteur_mediane
<chr> <chr> <dbl> <dbl>
1 94 VAL-DE-MARNE 296383 5.6
2 03 ALLIER 432031 4.3
3 76 SEINE-MARITIME 895789 4.5
4 53 MAYENNE 354627 4.6
5 36 INDRE 313658 4
# ℹ 91 more rowsA retenir
Arrowvous permet de lire très rapidement des jeux de données massifs et d’y appliquer des opérations d’agrégation simples.DuckDBvous permet de lire très rapidement des jeux de données massifs et d’y appliquer presque tout type d’opérationCes deux outils sont encore plus performants sur des jeux de données au format
Parquet, que l’on est amené à trouver de plus en plus souvent sur internet (Insee, datasoft, etc.)Seules adaptations nécessaires de votre code :
- Arrow : remplacer la lecture par une indexation :
open_dataset()à la place deread_XXX() - DuckDB : créer une connexion et y charger/indexer les données :
con <- dbConnect(duckdb());tbl(con, path)outbl_file(con, path)à la place deread_XXX(path) - Penser à “collecter” les données à la fin en format R si nécessaire :
collect()
- Arrow : remplacer la lecture par une indexation :
Qu’en est-il du spatial
Quelques packages en développement
| package | version CRAN | lifecycle | |
|---|---|---|---|
| duckspatial | R Interface to DuckDB Database with Spatial Extension (Cidre, 2025) | 0.2.0 | experimental |
| duckdbfs | duckdbfs: High Performance Remote File System, Database and ‘Geospatial’ Access Using ‘duckdb’ (Boettiger, 2025) | 0.1.2 | experimental |
| ducksf | ducksf. Spatial Ops Faster Than sf and geos. (Kotov, 2024) | / | / |
duckspatial: permet quelques opérations de “création de données” (buffer, centroides) et d’algèbre (intersection)duckdbfs: puissant mais uniquement de l’algèbre spatial, intersection avec de nombreux prédicats (within, disjoin, contains…)ducksf: conçu pour améliorer l’efficacité de certaines opérations de sf en passant temporairement en logique base de données
duckspatial
- Téléchargement du fichier
library(sf)
url_com <- "https://adresse.data.gouv.fr/data/contours-administratifs/2023/geojson/communes-100m.geojson"
# lecture avec sf
communes <- st_read(url_com, quiet = TRUE)
st_crs(communes)
# selection des communes d'un département
com_sel <- communes %>%
filter(departement == "90") %>%
left_join(resume_commune, by = c("code" = "COMMUNE"),
copy = TRUE) %>%
mutate(pct_logements = nb_logements / sum(nb_logements))- Jointure avec la table
resume_commune
- Passage en base de données avec duckspatial
duckspatial et SQL
Charger un csv et construire les géométries en SQL
# Télécharger le fichier CSV depuis le portail sncf
url_gares <- "https://ressources.data.sncf.com/api/explore/v2.1/catalog/datasets/gares-de-voyageurs/exports/csv?use_labels=true"
library(duckdb)
library(dplyr)
# Création de la table DuckDB directement à partir du CSV distant
dbExecute(con, paste0("
CREATE OR REPLACE TABLE gares_raw AS
SELECT * FROM read_csv_auto('", url_gares, "', delim=';', header=true)
"))- Mettre les données au propre et construire les géométries ponctuelles
# Transformation et création de la géométrie dans DuckDB
dbExecute(con, "
CREATE OR REPLACE TABLE gares_clean AS
SELECT
*,
CAST(SPLIT_PART(\"Position géographique\", ',', 1) AS DOUBLE PRECISION) AS lat,
CAST(SPLIT_PART(\"Position géographique\", ',', 2) AS DOUBLE PRECISION) AS lon,
FROM gares_raw
WHERE \"Position géographique\" IS NOT NULL
")- Créer la table finale avec la géométrie au format GEOMETRY
- Connaitre les tables et faire une intersection
Qu’en est-il du spatial
* Des packages qui semblent complementaires … mais pas d’interropérabilité entre packages (duckdb, duckspatial etc..)
* On finit toujours pas remettre les données en mémoire
* Finalement pas de package tout à fait consolidé, mais des avancées prometeuses notamment pour duckdbfs